BIOINFORMATIC KARACİĞER HASTALIĞI ANALİZİ VE TAHMİNİ

BIOINFORMATIC KARACİĞER HASTALIĞI ANALİZİ VE TAHMİNİ
Karaciğer hastalıkları dünya çapında önemli bir halk sağlığı sorunudur. Karaciğer, vücutta birçok temel işlevi yerine getiren en önemli organlardan biridir. Bu nedenle, karaciğer hastalıkları birçok ciddi sağlık sorununa neden olabilir. Karaciğer hastalıkları alkol tüketimi, hepatit enfeksiyonları, otoimmün hastalıklar ve genetik faktörler gibi çeşitli faktörlerden kaynaklanabilir.
Karaciğer hastalıklarında tanının doğru zamanda konulması çok önemlidir. Tanı ve tedavide geleneksel yöntemler yerine yapay zeka teknolojileri daha fazla kullanılıyor. Bu yapay zeka teknolojileri hastalığın öngörülmesinde çok faydalı olmaktadır.
Proje Kapsamı
Bu projenin amacı karaciğer hastalığı üzerine bir araştırma sunmaktır. Bu proje, karaciğer hastalığını tahmin etmek için kullanılan veri kümesini ve yapay zeka yöntemlerini içermektedir. Projedeki veri seti karaciğer hastalığını tahmin etmek için kullanılmaktadır.
Karaciğer Hastalığı Veri Seti
Bu veri seti, Hindistan’ın Andhra Pradesh eyaletinin Kuzey Doğusundan toplanan 416 karaciğer hastası kaydı ve 167 karaciğer hastası olmayan hasta kaydı içermektedir. (Şekil 1) “Veri Seti” sütunu, grupları karaciğer hastası (karaciğer hastalığı) veya değil (hastalık yok) olarak ayırmak için kullanılan bir sınıf etiketidir. Bu veri seti 441 erkek hasta kaydı ve 142 kadın hasta kaydı içermektedir. Veri setimizde 11 sütun bulunmaktadır (Şekil 2).
Gerekli Kütüphaneler
Proje Python dili ile yazılmıştır. 6 farklı Python kütüphanesi dahil edilmiştir. Bunlar NumPy, Pandas, Matplotlib, Seaborn, Warnings ve Os’dir.
NumPy bilimsel hesaplamalar için kullanılır ve matrisleri destekler.
Pandas veri analizi için bir kütüphanedir.
Matplotlib analiz işlemlerinde görselleştirme için kullanılır.
Seaborn matplotlib tabanını kullanır, yüksek seviye görselleştirme için kullanılır.
Uyarılar, uyarı mesajlarının filtrelenmesini sağlar.
Veri Kümesi Analizi
Veri setimizdeki veriler ile belirli analizler yapacağız.
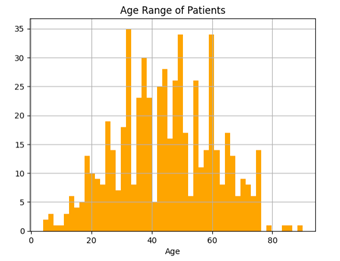
Şekil 3’e baktığımızda karaciğer hastalığının her yaş grubunda ortaya çıkabileceğini anlıyoruz. Ancak en yaygın yaş ortalamasının 30-60 olduğunu söyleyebiliriz.
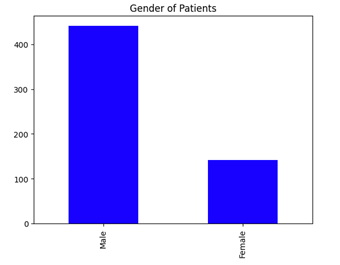
Şekil 4’e baktığımızda karaciğer hastalığının erkeklerde daha sık görüldüğünü söyleyebiliriz.
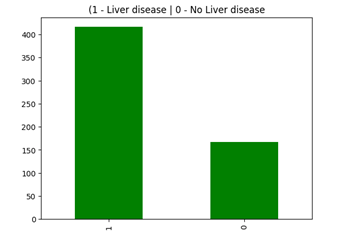
Veri setimizdeki verilere göre, Şekil 5’te karaciğer hastalığı olan 416 kişi ve karaciğer hastalığı olmayan 167 kişi bulunmaktadır.
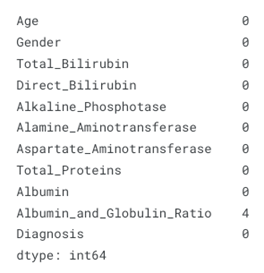
Şekil 6’ya baktığımızda, Albümin ve Globülin Oranının veri setimizde 4 boş değere sahip olduğunu görüyoruz. Bu boşluğu Albümin ve Globülin Oranının ortalamasını alarak dolduruyoruz. Eğer bunu yapmazsak, tahminimiz yanlış olabilir.
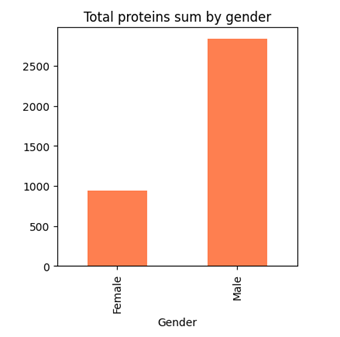
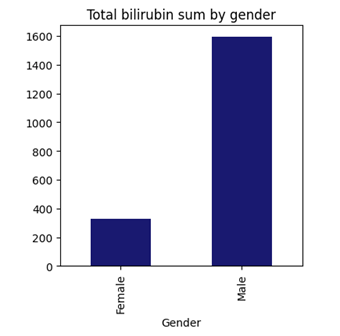
Şekil 7 ve 8’de Total protein ve Total bilirubinin erkeklerde yüksek olduğunu görüyoruz. Total bilirubin karaciğer hastalığı üzerinde en fazla etkiye sahiptir. Bilirubin normalde karaciğerde işlenerek vücuttan atılır, ancak karaciğerdeki bir sorun nedeniyle işlenemezse kanda birikir ve total bilirubin seviyesi yükselir. Bu yüksek seviyeler bazı karaciğer hastalıklarının belirtilerinden biridir.
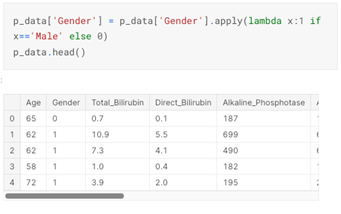
Şekil9’da cinsiyeti sayısal formata dönüştürüyoruz. 1= Erkek 0= Kadın.
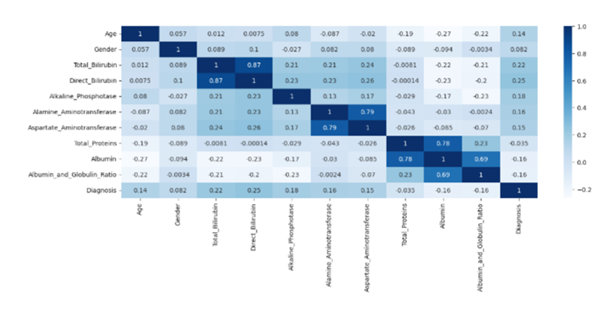
Şekil10’da değerleri görmek için heat meap kullanıyorum. Correlation_matrix kullanarak tablodaki veriler ile diğer veriler arasındaki ilişkileri ve benzerlikleri görüyoruz.
1 ile gösterilen yerler değerlerin kesişim noktalarıdır. Total_Bilirubin ve Direct_Bilirubin aynı değere sahiptir. Alamin ve Aspartat aynı değere sahiptir. Total_Proteins ve Albumin aynı değere sahiptir. Albümin ve Albümin Globülin Oranı aynı değere sahiptir.
SINIFLANDIRMA MODELI
Veri setimiz ile tahminler yapabilmemiz için öncelikle bu veri setini modelimize öğretmemiz gerekiyor.
Öncelikle teşhis sütununu veri setimizden ayırıyoruz ve corr yöntemini kullanarak diğer sütundaki verilerle ilişkisini belirliyoruz (Şekil 11).


Şekil 13’te ilk olarak StandardScaler sınıfını kullanarak skaler nesne oluşturdum, daha sonra X veri kümesindeki istatistiksel özellikleri fit yöntemi ile hesapladım.


LOGISTIC REGRESSION
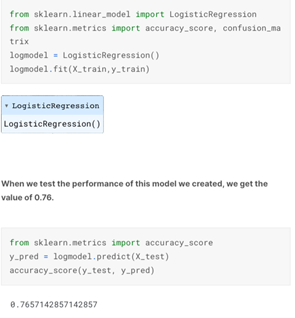
Logistic regresyon bir sınıflandırma yöntemidir ve bir şeyin iki kategoriden birine ait olup olmadığını tahmin etmek için kullanılır. Bu yöntemi karaciğer hastalığını tahmin etmek için de kullanacağız. Lojistik Regresyon sonucumuz 0.76’dır (Şekil 15)
SVC CLASSIFIER
SVC (Support Vector Classification) makine öğrenmesinde sınıflandırma problemleri için kullanılan bir algoritmadır. SVC yöntemi ile veri setimizin skorunun 0,77 olduğunu görüyoruz. Bu durumda tahminlemeyi SVC yöntemi ile yapacağım.

TAHMİN YÖNTEMİ
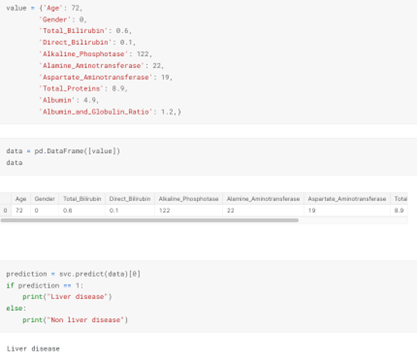
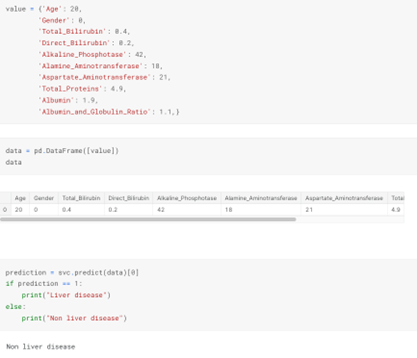
Sonuç olarak Şekil 16’da SVC yöntemi ile verdiğimiz değerlere göre hastamızda karaciğer hastalığı olduğu gözükmektedir. Şekil 17’de ise hastamızda karaciğer hastalığı bulunmuyor
import numpy as np
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
import warnings
import os
Liver Disease Analys
In this project I use liver_patient dataset. This dataset has 416 liver patient records and 167 non liver patient records. Also this data set contains 441 male patient records and 142 female patient records.
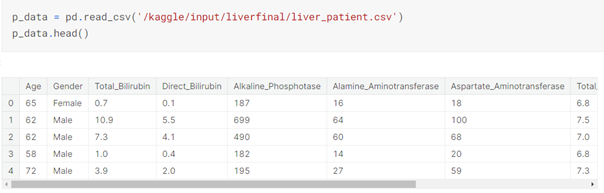
p_data = pd.read_csv('/kaggle/input/liverfinal/liver_patient.csv')
p_data.head()
Age Gender Total_Bilirubin Direct_Bilirubin Alkaline_Phosphotase Alamine_Aminotransferase Aspartate_Aminotransferase Total_Protiens Albumin Albumin_and_Globulin_Ratio Dataset
0 65 Female 0.7 0.1 187 16 18 6.8 3.3 0.90 1
1 62 Male 10.9 5.5 699 64 100 7.5 3.2 0.74 1
2 62 Male 7.3 4.1 490 60 68 7.0 3.3 0.89 1
3 58 Male 1.0 0.4 182 14 20 6.8 3.4 1.00 1
4 72 Male 3.9 2.0 195 27 59 7.3 2.4 0.40 1
p_data.shape
(583, 11)
We have 583 row and 11 colums in dataset
> DATA ANALYSIS
p_data['Age'].hist(bins=50, color='orange')
plt.title("Age Range of Patients")
plt.xlabel("Age");
p_data['Gender'].value_counts().plot.bar(color='blue')
plt.title("Gender of Patients");
In this dataset we has dataset column. Dataset column mean have liver disease or non liver disease
p_data.rename(columns={'Dataset': 'Diagnosis', 'Total_Protiens': 'Total_Proteins'}, inplace=True)
p_data['Diagnosis'] = p_data['Diagnosis'].apply(lambda x:1 if x==1 else 0)
p_data['Diagnosis'].value_counts().plot.bar(color='green')
plt.title('(1 - Liver disease | 0 - No Liver disease');
p_data.isnull().sum()
Age 0
Gender 0
Total_Bilirubin 0
Direct_Bilirubin 0
Alkaline_Phosphotase 0
Alamine_Aminotransferase 0
Aspartate_Aminotransferase 0
Total_Proteins 0
Albumin 0
Albumin_and_Globulin_Ratio 4
Diagnosis 0
dtype: int64
average_ag= p_data['Albumin_and_Globulin_Ratio'].mean()
average_ag
0.9470639032815197
p_data = p_data.fillna(average_ag)
p_data.isnull().sum()
Age 0
Gender 0
Total_Bilirubin 0
Direct_Bilirubin 0
Alkaline_Phosphotase 0
Alamine_Aminotransferase 0
Aspartate_Aminotransferase 0
Total_Proteins 0
Albumin 0
Albumin_and_Globulin_Ratio 0
Diagnosis 0
dtype: int64
plt.figure(figsize=(4,4))
p_data.groupby('Gender').sum()["Total_Proteins"].plot.bar(color='coral')
plt.title('Total proteins sum by gender');
Protein intake is higher in male
plt.figure(figsize=(4,4))
p_data.groupby('Gender').sum()['Total_Bilirubin'].plot.bar(color='midnightblue')
plt.title('Total bilirubin sum by gender');
p_data['Gender'] = p_data['Gender'].apply(lambda x:1 if x=='Male' else 0)
p_data.head()
Age Gender Total_Bilirubin Direct_Bilirubin Alkaline_Phosphotase Alamine_Aminotransferase Aspartate_Aminotransferase Total_Proteins Albumin Albumin_and_Globulin_Ratio Diagnosis
0 65 0 0.7 0.1 187 16 18 6.8 3.3 0.90 1
1 62 1 10.9 5.5 699 64 100 7.5 3.2 0.74 1
2 62 1 7.3 4.1 490 60 68 7.0 3.3 0.89 1
3 58 1 1.0 0.4 182 14 20 6.8 3.4 1.00 1
4 72 1 3.9 2.0 195 27 59 7.3 2.4 0.40 1
We convert the gender to numeric format. 1= Male 0= Female
corr=p_data.corr()
plt.figure(figsize=(15,5))
sns.heatmap(corr,cmap="Blues",annot=True)
<AxesSubplot: >
CREATE CLASSIFICATION MODEL
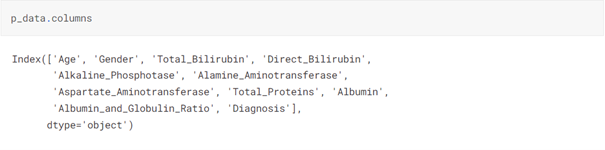
p_data.columns
Index(['Age', 'Gender', 'Total_Bilirubin', 'Direct_Bilirubin',
'Alkaline_Phosphotase', 'Alamine_Aminotransferase',
'Aspartate_Aminotransferase', 'Total_Proteins', 'Albumin',
'Albumin_and_Globulin_Ratio', 'Diagnosis'],
dtype='object')
p_data.drop("Diagnosis", axis=1).apply(lambda x: x.corr(p_data['Diagnosis']))
Age 0.137351
Gender 0.082416
Total_Bilirubin 0.220208
Direct_Bilirubin 0.246046
Alkaline_Phosphotase 0.184866
Alamine_Aminotransferase 0.163416
Aspartate_Aminotransferase 0.151934
Total_Proteins -0.035008
Albumin -0.161388
Albumin_and_Globulin_Ratio -0.162319
dtype: float64
# x is main data , y is our feature data
X = p_data.drop('Diagnosis', axis=1)
y = p_data['Diagnosis']
from sklearn.preprocessing import StandardScaler
scaler = StandardScaler().fit(X)
X_scaled = scaler.transform(X)
from sklearn.model_selection import train_test_split
X_train,X_test,y_train,y_test=train_test_split(X,y,test_size=0.3,random_state= 21)
LOGISTIC REGRESSION
from sklearn.linear_model import LogisticRegression
from sklearn.metrics import accuracy_score, confusion_matrix
logmodel = LogisticRegression()
logmodel.fit(X_train,y_train)
LogisticRegression
LogisticRegression()
When we test the performance of this model we created, we get the value of 0.76.
from sklearn.metrics import accuracy_score
y_pred = logmodel.predict(X_test)
accuracy_score(y_test, y_pred)
0.7657142857142857
from sklearn.metrics import classification_report
print(classification_report(y_test, y_pred))
precision recall f1-score support
0 0.45 0.34 0.39 38
1 0.83 0.88 0.86 137
accuracy 0.77 175
macro avg 0.64 0.61 0.62 175
weighted avg 0.75 0.77 0.75 175
SVC Classifier
from sklearn.svm import SVC
svc = SVC(kernel='linear')
svc.fit(X_train, y_train)
y_pred = svc.predict(X_test)
accuracy_score(y_test, y_pred)
0.7771428571428571
print(classification_report(y_test, y_pred))
precision recall f1-score support
0 0.00 0.00 0.00 38
1 0.78 0.99 0.87 137
accuracy 0.78 175
macro avg 0.39 0.50 0.44 175
weighted avg 0.61 0.78 0.68 175
value = {'Age': 72,
'Gender': 0,
'Total_Bilirubin': 0.6,
'Direct_Bilirubin': 0.1,
'Alkaline_Phosphotase': 122,
'Alamine_Aminotransferase': 22,
'Aspartate_Aminotransferase': 19,
'Total_Proteins': 8.9,
'Albumin': 4.9,
'Albumin_and_Globulin_Ratio': 1.2,}
data = pd.DataFrame([value])
data
Age Gender Total_Bilirubin Direct_Bilirubin Alkaline_Phosphotase Alamine_Aminotransferase Aspartate_Aminotransferase Total_Proteins Albumin Albumin_and_Globulin_Ratio
0 72 0 0.6 0.1 122 22 19 8.9 4.9 1.2
prediction = svc.predict(data)[0]
if prediction == 1:
print("Liver disease")
else:
print("Non liver disease")
Liver disease
value = {'Age': 20,
'Gender': 0,
'Total_Bilirubin': 0.4,
'Direct_Bilirubin': 0.2,
'Alkaline_Phosphotase': 42,
'Alamine_Aminotransferase': 18,
'Aspartate_Aminotransferase': 21,
'Total_Proteins': 4.9,
'Albumin': 1.9,
'Albumin_and_Globulin_Ratio': 1.1,}
data = pd.DataFrame([value])
data
Age Gender Total_Bilirubin Direct_Bilirubin Alkaline_Phosphotase Alamine_Aminotransferase Aspartate_Aminotransferase Total_Proteins Albumin Albumin_and_Globulin_Ratio
0 20 0 0.4 0.2 42 18 21 4.9 1.9 1.1
prediction = svc.predict(data)[0]
if prediction == 1:
print("Liver disease")
else:
print("Non liver disease")
Non liver disease